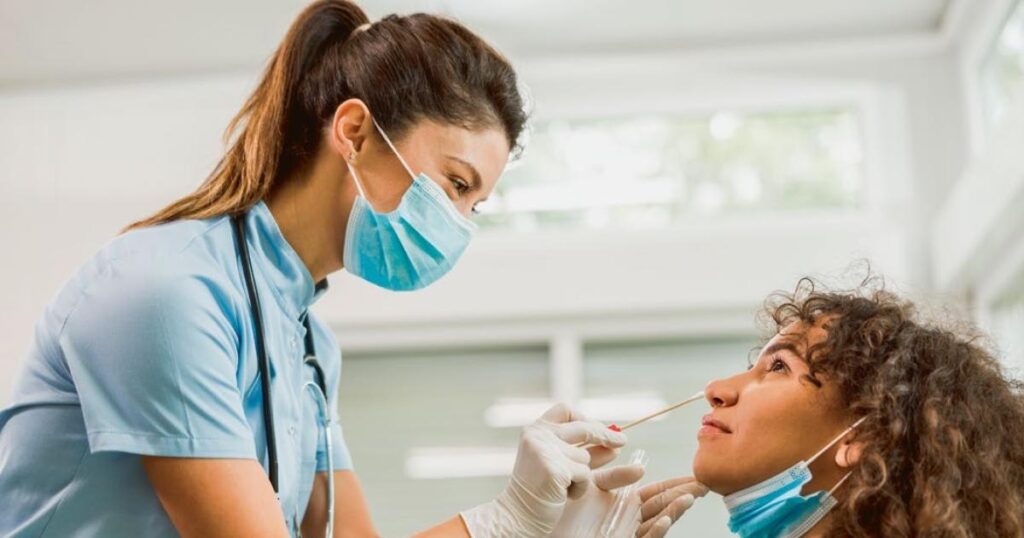
La crisis pandémica ha acercado al dominio público términos y conceptos hasta ahora exclusivos del ámbito científico-técnico. Uno de ellos es la PCR, iniciales de polymerase chain reaction («reacción en cadena de la polimerasa»). Pero la PCR es mucho más que una técnica diagnóstica aplicada a muestras tomadas mediante el incómodo exudado nasofaríngeo para detectar el maldito SARS-CoV-2. La PCR, que le valió el Nobel a su controvertido descubridor, Kary Mullis (1944-2019), ha catalizado una reacción en cadena (valga la redundancia) de avances experimentales desde la década de 1990, una revolución asombrosa en el ámbito de la genética y la biología molecular y celular. Ha permitido el nacimiento y aceleración de la genómica y la posgenómica, el auge de la biotecnología y el desarrollo de técnicas moleculares que hoy son rutina imprescindible en múltiples campos.
Desde 1991, la PCR se ha convertido en una herramienta imprescindible para amplificar y modificar ADN in vitro en los laboratorios.
Clonación en laboratorio
La PCR es una técnica que permite sintetizar grandes cantidades de una determinada secuencia de ADN in vitro. En el laboratorio, hablamos de «amplificar» un fragmento de ADN, porque la secuencia acotada por nuestro diseño experimental permite, a partir de una sola molécula, obtener más de mil millones de copias idénticas en pocas horas… Incluso en menos de una hora si usamos la tecnología más moderna. Hasta su desarrollo, la única manera de obtener suficiente cantidad de ADN para realizar procesos de clonación en el laboratorio de biología molecular era mediante protocolos de fraccionamiento químico a partir de muestras biológicas. Hablamos de microgramos de ADN, que, en términos moleculares, es una cantidad enorme. No olvidemos que los biólogos moleculares trabajan en pequeños tubos, con micropipetas que funcionan en una escala de microlitros.
La herramienta biológica para manejar ADN recombinante era —sigue siendo y será— la bacteria Escherichia coli, un habitante de nuestro intestino (en concreto del colon, de ahí el nombre de la especie) domesticado por los investigadores hasta convertirlo en la fuente de material genético esencial en cualquier experimento de ingeniería genética. La introducción de pequeñas moléculas de ADN circulares, llamadas plásmidos, capaces de replicarse autónomamente en la bacteria mediante un proceso sencillo llamado «transformación», y la extracción de estos plásmidos a partir de cultivos de E. coli, mediante un protocolo de fraccionamiento químico conocido como «lisis alcalina», permitía la obtención de suficiente ADN para su análisis mediante separación electroforética en geles de agarosa y su manipulación en el laboratorio.
En 1983, cuando Mullis tuvo la iluminación que sentaría las bases de la PCR, la «ingeniería genética» había despertado ya gracias al desarrollo de reacciones in vitro que permitían cortar y pegar fragmentos de ADN utilizando como «tijeras» enzimas bacterianas purificadas (las llamadas endonucleasas de restricción) y una enzima de un virus bacteriófago (la ligasa de ADN del fago T4) como «pegamento». Paul Berg, pionero de estas estrategias y, por tanto, del nacimiento de la clonación molecular, recibió el Nobel en 1980, compartido con los padres de la secuenciación del ADN, Walter Gilbert y Fred Sanger.
Precisamente uno de los grandes retos de la biología molecular en esa época era la síntesis de ADN —la replicación de la doble hebra— in vitro. Se disponía de la enzima ADN polimerasa purificada, responsable de realizar la copia. De hecho, las técnicas de secuenciación por las que el jurado del Nobel premió a Gilbert y Sanger se basan en el uso de esta enzima. Sin embargo, sintetizar de manera eficaz grandes cantidades de ADN in vitro era inviable por un motivo concreto: para copiar la doble cadena hay que abrirla, separar ambas hebras, de manera que la polimerasa reconozca la cadena sencilla abierta, «lea» la secuencia y sintetice, base a base, la cadena complementaria, cerrando de nuevo la doble hélice. El proceso es similar a una cremallera que va cerrándose según crea una de las partes a partir de la otra. En las células, este proceso requiere el apoyo de una pléyade de proteínas accesorias que abren la hélice (helicasa), estabilizan la cadena sencilla (proteínas SSB, de single strand-binding) y permiten el avance bidireccional de la ADN polimerasa en la llamada «horquilla de replicación»: un proceso de enorme complejidad para reproducir el fenómeno in vitro, incluso hoy.
Controvertido e impredecible por sus ideas y sus costumbres, Kary Mullis, descubridor de la PCR y ganador del premio Nobel, murió condenado al ostracismo por sus compañeros de profesión.
Una idea al volante
Kary Mullis contribuyó a su propia leyenda relatando de manera novelada en un artículo para Scientific American (1990; volumen 262, páginas 56-65) y en sus memorias, Dancing Naked in the Mind Field (1998), el momento eureka en que dio con un plan B para abrir la doble hélice: desnaturalizarla a altas temperaturas. Los enlaces de hidrógeno que mantienen unidos los pares complementarios en la doble hélice, adenina-timina (A-T) y citosina-guanina (C-G), se relajan a temperaturas superiores a 90 ºC. Aparentemente, ocurrió mientras conducía por la pintoresca Highway 101 desde la bahía de San Francisco a su finca en el recoleto condado costero de Mendocino a través de los hermosos valles del norte de California y sus sorprendentes bosques de secuoyas. Mullis, que trabajaba para la biotecnológica Cetus Corporation (Emeryville, California), iba al volante dándole vueltas a estrategias para mejorar protocolos de detección de mutaciones puntuales (los llamados polimorfismos de un solo nucleótido o SNP, una de las claves de nuestra variabilidad genética) mediante síntesis de ADN in vitro cuando cayó en la cuenta de que sucesivos ciclos de calentamiento a temperatura de desnaturalización y enfriamiento a temperatura de síntesis podrían generar copias de un fragmento acotado que se duplicarían en cada ciclo, creciendo de manera exponencial, es decir, obteniendo 2n copias de la cadena acotada al cabo de n ciclos.
La acotación del fragmento elegido, lo que ahora llamamos «amplicón», vendría determinada por los denominados «primers». La ADN polimerasa no copia ADN de la nada, necesita continuar una cadena incompleta que, en la hebra discontinua de la horquilla de replicación en la célula, le proporciona otra enzima, denominada «primasa». Por tanto, es necesario añadir a la reacción oligonucleótidos (esos «primers», «cebadores» u «oligos» para los amigos), pequeños fragmentos de ADN monocatenario que, en direcciones opuestas, señalen los límites de la secuencia a amplificar. En la mezcla de reacción, además, por supuesto, debe estar el ADN molde, al menos una molécula, los cuatros desoxinucleótidos-trifosfato (dATP, dTTP, dGTP y dCTP), que son las «unidades» del código genético a encadenar por la polimerasa según el dictado del molde, así como las sales y tampones de pH óptimos para la reacción. Uno de los alicientes de Kary Mullis y de Cetus Corporation es que la compañía se dedicaba a sintetizar químicamente oligonucleótidos y el desarrollo de técnicas basadas en el uso de estos podía dar una salida comercial a sus productos.
Solo hay un pequeño problema: las proteínas son mucho más sensibles a las altas temperaturas que los ácidos nucleicos. Pierden su estructura de manera irreversible y se desnaturalizan e inactivan a temperaturas mucho más bajas. Malas noticias, claro: la ADN polimerasa purificada de E. coli que debe hacer el trabajo, como todas las enzimas, es una proteína. A temperaturas próximas a la ebullición, necesarias para abrir la doble hélice, la polimerasa está irremediablemente muerta. Esto no hace del todo inviable la idea de Mullis, pero sí tediosa y cara, pues implica inyectar ADN polimerasa nueva al enfriar en cada ciclo, en cuanto la temperatura bajase lo suficiente para garantizar la hibridación de los «primers» en sus secuencias complementarias diana en el molde y a temperaturas que no comprometiesen su actividad. Teniendo en cuenta que se necesitan treinta ciclos en una PCR, ello implica treinta adiciones de enzima.
Aun así, el equipo de científicos que trabajaba con Mullis en Cetus consiguió probar que la amplificación de ADN por esta reacción en cadena de la polimerasa era viable. El equipo publicó en Science en 1985 una aplicación del método aplicado al diagnóstico de la anemia falciforme (volumen 230, páginas 1350-1354), aunque la técnica en sí, con todo su potencial, no se publicaría hasta dos años después, cuando Mullis, excéntrico e impredecible, ya había abandonado la compañía.
La solución definitiva para que la PCR fuera una realidad viable como técnica universal vendría una vez más de la mano de la microbiología. Parece ser que el propio Kary Mullis, antes de abandonar la compañía, sugeriría a sus compañeros de equipo utilizar polimerasas de microorganismos termófilos a fin de simplificar el proceso. Fuera como fuera, su compañero David Gelfand concertó una visita al laboratorio del veterano microbiólogo Thomas Brock (1926-2021). Autor de uno de los libros de texto imprescindibles para los estudiantes de Microbiología durante décadas, a Brock se le considera una figura clave en el estudio de los extremófilos, las formas de vida que proliferan en condiciones límite. Su trabajo ha tenido una enorme influencia en la ecología microbiana y la microbiología medioambiental durante décadas.
En uno de sus artículos más citados e influyentes, «Life at high temperatures», en Science (1985; volumen 230, páginas 132-138), subraya el enorme potencial biotecnológico de las enzimas procedentes de microorganismos extremófilos. Su lectura pudo inspirar a Mullis y su equipo la idea de trabajar con una ADN polimerasa termorresistente, capaz de aguantar sin desnaturalizarse ni inactivarse las temperaturas necesarias para abrir la cremallera de la hélice de ADN. Si fuera lo suficientemente robusta, esta enzima permitiría automatizar los ciclos de PCR sin necesidad de parar y añadir la enzima en cada uno de ellos. Una bacteria termófila traería la solución: Thermus aquaticus.
Incómoda por la forma de obtener las muestras, la PCR no solo ha contribuido a la detección de la covid-19, sino que ha supuesto una revolución en distintos ámbitos de la ciencia.
Fuente de incógnitas
De nuevo al volante por los extensos paisajes norteamericanos y su sorprendente belleza natural, pero casi veinte años antes que Mullis, en 1964, Brock se desvió de la Highway 90 en su periplo de tres mil setecientos kilómetros desde su laboratorio en Indiana a Seattle, donde iba a realizar trabajos de campo en la Universidad de Washington, para hacer una visita al Parque Nacional de Yellowstone, en el estado de Wyoming. Los tapices de colores que los microorganismos dibujan en las aguas termales sulfurosas de los lagos de Yellowstone son una fotogénica maravilla natural para el visitante, pero para un microbiólogo son una fuente inagotable de incógnitas. La fisiología y la bioquímica de los seres que allí proliferaban, probablemente similar a la de las células primigenias que surgieron hace más de cuatro mil millones de años asociadas a aguas volcánicas en un planeta aún sin oxígeno, era un reto fascinante.
Obsesionado por la biología de estos microorganismos capaces de vivir a temperaturas cercanas a la ebullición, Brock regresó en sucesivas ocasiones con la idea de tomar muestras de los tapetes microbianos de Yellowstone e intentar cultivar en su laboratorio estas formas de vida fascinantes. En 1969, junto con su colega Hudson Freeze, publicaría el aislamiento de una bacteria de forma bacilar cuya temperatura óptima de crecimiento está entre 65 y 70 ºC (J. Bacteriol, volumen 98, páginas 289-297): Thermus aquaticus. Siete años más tarde se purificaría la ADN polimerasa de esta bacteria, conocida con el acrónimo de Taq polimerasa. Su temperatura óptima de trabajo es de 72 ºC y sintetiza ADN a una velocidad de unos cincuenta nucleótidos por segundo, pero más importante aún es su termoestabilidad: se mantiene intacta hasta 95 ºC.
Es la pieza ideal que faltaba para componer el rompecabezas de la PCR: una sola adición de Taq al principio del proceso, junto con suficiente cantidad de dNTP y «primers», sería suficiente para amplificar doscientas treinta veces la secuencia diana en treinta ciclos, de un tirón, en poco más de una hora. El descubrimiento se publicaría en 1988 (Science, volumen 239, páginas 487-491) y lo demás es historia: el desarrollo y patente de los primeros «termocicladores», aparatos diseñados para albergar los tubos de reacción y programar de forma cíclica con la máxima eficiencia temperaturas consecutivas de 95 ºC (desnaturalización del ADN molde), 55 oºC (hibridación de los «primers») y 72 ºC (síntesis del ADN por la Taq), terminó con la venta de derechos de la PCR a la corporación Roche por trescientos treinta millones de dólares en 1991. En cuestión de meses, todos los laboratorios de biología molecular del mundo tendrían un termociclador y obtener una buena cantidad de cualquier gen para su clonación molecular y otras técnicas de ADN recombinante dejó de ser un obstáculo para el avance de la Biomedicina y la Biotecnología.
En 1964, Thomas Brock visitó el Parque Nacional de Yellowstone para tomar muestras de los tapetes microbianos e intentar cultivarlos en su propio laboratorio.
Manipulación genética in vitro
Desde 1991, la PCR es la herramienta imprescindible para amplificar y modificar ADN in vitro en nuestros laboratorios. Permite obtener a partir de cualquier tipo de muestra el gen que deseemos para su expresión en cualquier sistema, facilitando el desarrollo de múltiples herramientas. Utilizando «primers» con secuencias adicionales en sus colas 5’ (para que un «primer» funcione basta con que las quince últimas bases en su región 3’ sean idénticas por complementariedad de bases A-T G-C a la secuencia diana que se desea amplificar) se pueden introducir dianas de enzimas de restricción para clonaje tradicional, generar quimeras, fusiones génicas, construcciones génicas para realizar sustituciones alélicas por recombinación, etcétera.
Se puede utilizar también para realizar mutagénesis en el laboratorio tanto al azar, disminuyendo la enorme fidelidad de copia de la Taq polimerasa para que cometa errores, como de manera dirigida, incluyendo «mismatches» en los «primers» que introduzcan la mutación deseada de la secuencia diana. Las posibilidades para la manipulación génica in vitro de la ADN mediante PCR son ilimitadas. La secuenciación de genomas, el hito científico del milenio, tanto de organismos modelo en biomedicina como la del subsiguiente genoma humano, no se habrían conseguido fácilmente sin ayuda de la PCR. Se ha convertido en la técnica de diagnóstico molecular de patologías genéticas y de todo tipo de patógenos.
Noticia: ARN mensajero
La realización de un paso previo de transcripción reversa (copia de ARN a ADN) sobre muestras clínicas permite la detección y amplificación de virus cuyo genoma es ARN, como es el caso del VIH y de los coronavirus. Mejoras tecnológicas como la PCR cuantitativa a tiempo real (qrtPCR) y la PCR digital (dPCR) hacen posible no solo detectar, sino aproximar la cantidad de moléculas de ácido nucleico en la muestra, lo que es muy útil en determinación de carga viral y en estudios básicos de expresión génica en células o tejidos.
La identificación de especies microbianas se basa en la amplificación por PCR de un fragmento de los genes que codifican el ARN ribosomal 16S, una secuencia que funciona a modo de carnet de identidad de las especies microbianas y nos ayuda a clasificarlas utilizando su distancia evolutiva como criterio.
Las huellas genéticas que se utilizan en pruebas judiciales para incriminación de sospechosos, paternidad y las que se diseñan para estudiar clonalidad en patógenos con fines epidemiológicos para investigar brotes de infecciones, como por ejemplo en casos de listeriosis y salmonelosis, también se han beneficiado de la PCR. Sus aplicaciones son simplemente innumerables. La PCR está en la base de la ciencia y la tecnología en la investigación de frontera en las determinadas «-ómicas» y en el desarrollo de todo tipo de herramientas biotecnológicas de última generación. La propia secuenciación de alto rendimiento, que permite hoy en día secuenciar un genoma humano completo en horas, se basa técnicamente en miniaturización y automatización de la PCR.
Hay dos propiedades esenciales que justifican el éxito de la Taq polimerasa: su fidelidad y su procesividad. La primera implica que no se equivoque nunca al copiar la cadena molde, porque entonces introduciría mutaciones, errores en la secuencia, «ruido», lo cual es indeseable en todos los casos salvo en experimentos de mutagénesis al azar. La segunda es su capacidad de sintetizar secuencias de miles de bases en cada ciclo sin agotarse. Dado el lucrativo mercado de la PCR, las compañías biotecnológicas han desarrollado durante las últimas décadas polimerasas mejoradas para PCR, no solo mejorando la propia Taq, sino lanzando polimerasas procedentes de otros termófilos. La Pfu, por ejemplo, es una polimerasa aislada de Pyrococcus furiosus, una arquea hipertermófila, como indica su nombre de forma explícita, con una temperatura óptima de crecimiento de 100 ºC. Las arqueas son procariotas pertenecientes a un dominio distinto a las bacterias que son vestigiales de la vida en una tierra primitiva y colonizan ambientes extremos.
La proteína Sso7d procede de una arquea termoacidófila, Sulfolobus solfataricus, que fue aislada
en 1980 en el volcán Solfatara, en la Campania italiana.
Un origen volcánico
En concreto, P. furiosus se encontró en sedimentos marinos volcánicos en una isla del Mediterráneo. La presión que ejerce la columna de agua en los fondos marinos aumenta su temperatura de ebullición y permite la vida de este tipo de organismos en estas fuentes termales volcánicas submarinas. La Pfu es más procesiva que la Taq y se ha conseguido incluso aumentar dicha procesividad manteniendo una alta fidelidad, generando una quimera con una proteína rica en cargas positivas (Sso7d), lo que, al igual que las proteínas que constituyen nuestra cromatina, facilita su unión a ADN. Dicha proteína, por supuesto, procede también de una arquea termoacidófila, Sulfolobus solfataricus, aislada en 1980 en el volcán Solfatara, en la Campania italiana, cuya temperatura óptima de crecimiento es de 80 ºC. Una elevada procesividad acorta notablemente el tiempo necesario en cada ciclo y permite obtener amplicones mayores. La Pfu y los híbridos Pfu-Sso7d amplifican secuencias de ADN de hasta diecinueve mil pares de bases, mientras que el límite de procesividad de la Taq está en torno a cinco mil pares de bases.
Mullis falleció en 2019 condenado al ostracismo intelectual por el grueso de la comunidad científica. Consumidor confeso de LSD y otras drogas, tras el Nobel, que según sus colegas se le subió a la cabeza, se dedicó al surf, a dar conferencias no exentas de comentarios sexistas, a supuestos encuentros con extraterrestres y mapaches fluorescentes y a negar públicamente la existencia del cambio climático, el agujero de ozono y el HIV. De haber vivido para conocer la covid-19, probablemente habría abrazado también el negacionismo, al tiempo que, paradójicamente, habría visto su invención traspasar la frontera de los laboratorios hacia la sociedad en los campos del diagnóstico clínico y la epidemiología. Podemos repudiar a Mullis sin negarle el genio de la autoría del descubrimiento. Es un ejemplo paradigmático de cómo las ideas útiles más brillantes se fundamentan siempre sobre una sólida base de conocimiento básico. En este caso, además de a la inspiración de Mullis, se lo debemos a décadas de estudios sobre biología molecular producto de estudios realizados en E. coli y, sobre todo, a las fascinantes bacterias termófilas.
Fuente de TenemosNoticias.com: www.muyinteresante.com
Publicado el: 2024-06-29 12:30:00
En la sección: Muy Interesante